2020.09.21
新型コロナウィルス感染症の特徴は感染から発症までの時間遅れ(潜伏期間)と発症前における感染力である。この原因としては、ウィルスの持つ自然免疫妨害機能があるらしいが、ともあれ、その為に、未発症状態での感染が多く、制御が難しい。そういう意味で、感染力についての論文を調べた。方法としては二つあって、一つは患者の排出するウィルス量を調べる方法である。これはPCR検査でRNAが見つかると、その検体を培養してウィルスが増殖するかどうか、つまり生きたウィルスが居るかどうかを調べる。もう一つは、実際に感染をさせた側(一次感染者)と感染を受けた側(二次感染者)の対を同定して、数多く集めてその統計から感染力の分布を得る方法である。更には、これらの研究論文を Review した論文も多数ある。殆どの研究は軽症の発症者についてのものであるが、未発症のまま終わる患者(不顕性感染者)もかなりの数であることが推定されていて、その割合や感染力についての論文も重要である。何故なら、不顕性感染者については全員検査しない限り発見が難しいので、感染制御を更に難しくするからである。以下おおむね読んだ順に並んでいる。記号の定義が各論文によって異なるので注意してほしい。なお、モデル計算の論文については別途まとめる。後日(12月24日)訂正補足箇所の主要部を赤字 で示した。
9月4日
K. Mizumoto et al., "Estimating the asymptomatic proportion of coronavirus disieas 2019 (COVID-19) cases on board the Diamond Princess cruise ship, Yokohama, Japan, 2020"
https://www.eurosurveillance.org/content/10.2807/1560-7917.ES.2020.25.10.2000180
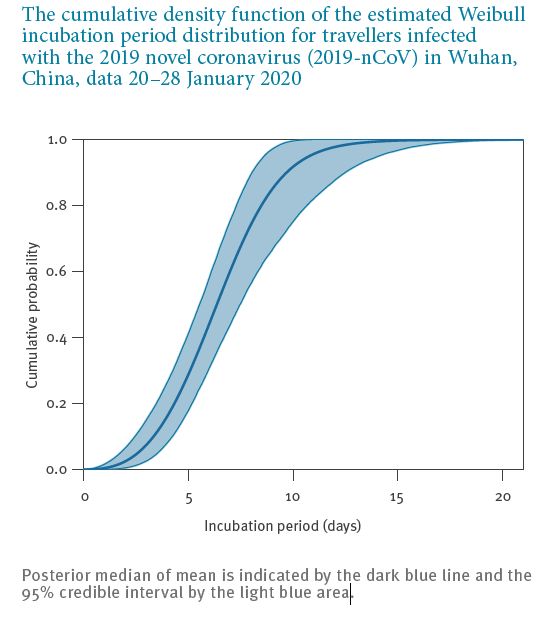
使ったデータは、ダイアモンドプリンセス号における、2020年、5月2日〜20日までのPCR検査データ、感染が起きたと思われる日付(x)、その時(c)発症していたかどうか、である。基本的なモデルは、未発症のままで治癒してしまう感染者(不顕性感染者)の割合 p 、それ以外、(1−p) の割合で発症する場合 の発症日の分布には観測値を使う。平均発症日は感染から 6.4 日後で、標準偏差 2.3 日の Weibull 分布である。これを FD(c−x) と表記する。累積分布、不顕性でない患者が感染から c−x 日経過したときに発症している確率である。下記論文のデータを使っている。
J.A.Backer et al.,"Incubation period of 2019 novel coronavirus (2019-nCoV) infections among travellers from Wohan, China, 20-28 Jan. 2020". Euro Surveill. 2020;25(5).https://doi.org/10.2807/1560-7917.
ES.2020.25.5.2000062 PMID:32046819
測定日に発症していれば、確実に 1−p の組に入る。発症していなければ、まだ発症していないだけなのかもしれないので、それは
確率 g(x,p)=p+(1−p)(1−FD(c−x)) が実現している。つまり、不顕性感染者である確率+未発症である確率。
もしも発症していれば、それは 確率 g(x,p)=(1−p)FD(c−x) が実現している。これは、発症を死亡に置き換えれば、「生存解析」と同じである。ベイズ法で計算したらしいが、詳細は勉強していないので判らない。結果は 95%信頼区間で、0.155<p<0.202、平均値(最終的な生存率に相当する)は 0.179 である。
この段階では、未発症感染者が感染能力があるとは確認されていなかったが、他の伝染病ではよくあることだから、あり得るという議論がなされている。
幾つかの制約条件がある。検査には優先順位があったので、ランダムサンプリングではない。高齢者が多いので、不顕性感染者は少なめになっているだろう。
僕の印象としては、折角感染者を全て隔離して観察したのに、その結果を使っていないのは何故なのだろう、という疑問である。つまり、統計的手法を使わなくても、その後発症したかどうかが判っているのではないか?多分データの使用が許されなかったのだろう。また、ここでは、発症日分布関数 FD(c−x) が重要な役割を果たしている。その信頼性はどうか?いずれにしても、2週間ばかりの間に治癒してしまう感染者は居なかったのだろうか?発症前で2週間以内に治癒した場合は、不顕性感染者に算入するのだろうか? おそらく、高齢者なので、居なかったのだろう。。。
Xi He et al. , Temporal dynamics in viral shedding and transmissibility of COVID-19, Nat Med 26, 672–675 (2020), https://doi.org/10.1038/s41591-020-0869-5 これは Nature の Free Access になっているのだが、ダウンロードしようとすると途中で止まってしまう。仕方ないので、MedRxiv にある Preprint を読んだ。(後日、最終稿を読んだが、結果に変更があるので注意。重要な infectoiusness profile が更新されている 。)
viral shedding というのは ウィルス排出、という意味である。PCR検査で検出されるウィルス量のことである。
この論文は非常に有名で、COVID-19 が発症前から感染を拡げている、ということを明確に示して、感染対策に大きな影響を与えた。発症と感染性の時間関係を推定している。ベースとなる論文は W. Peng et al., Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia,
New England Journal of Medicine · January 2020 DOI: 10.1056/NEJMoa2001316
であるが、引用されているのは、筆頭が Q. Li になっている。その論文では、感染から発症に至る日数の分布関数がまとめられている。そういう研究は他にも非常に多いが、同じ研究室の結果を使ったということであろう。分布関数を Inc(t) としておく。incubation period ditribution と呼ぶ。
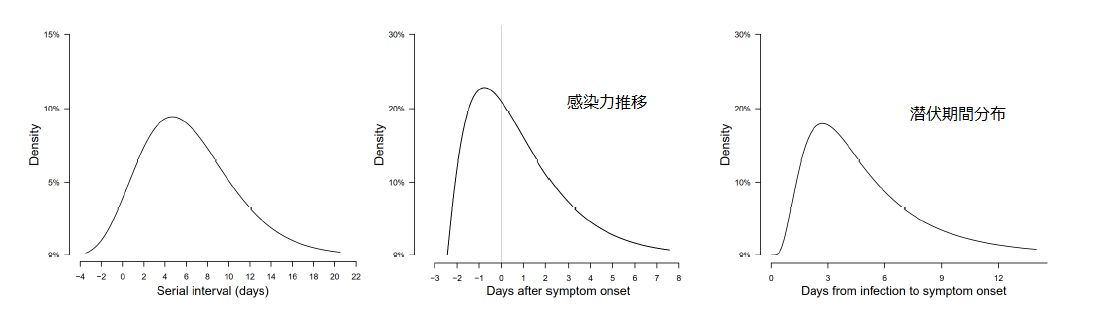
重要なデータは、感染者と被感染者の対のデータである。多くのデータから純粋にその2人の間で感染が起きた事の明確なものを 74 例選んでいる。その上で、2人の発症日の差を採り上げて、その分布関数を作る。これを f(t) とする。serial interval distribution と呼んでいる。そこで、発症日を基準として、感染性がどれくらいに推移するか、という分布関数を考える。これを Inf(t) とする。infectiousness profile と呼んでいる。
これら3つの分布関数は、次の式で繋がっている。
f(ts2−ts1)=∫Inc(ts2−t1)Inf(t1−ts1)dt1
最初の人が ts1 で発症したとすると、その人が t1 において持つ感染力は Inf(t1−ts1) である。これは次の人を t1 において感染させる確率に比例する。そこで、t1 で感染したとして、感染した人が ts2 に発症する確率は、Inc(ts2−t1) である。従って、考え得る t1 について積分すれば、最初の人が ts1 において発症したという条件の元で、2番目の人が ts2 において発症する確率は、上の式のようになる。
こうして、2つの知られた分布関数から未知の分布関数 Inf を推定する、という数学的問題に帰着される。関数形さえ決めれば、統計的手法で決めることができる。こういう場合には、Maximum likelyhood method が開発されていて、R を使うのが一般的になっている。(Rを使えない人はデータが扱えないから論文が書けない、とも言える。。。僕はもう忘れてしまった。。。)関数形を推定する上で重要なのが、PCR検査である。多数の患者のPCR検査結果(ウィルス量)を発症日基準で整理すると、発症日からのなだらかな減少関数となる。ウィルス量は感染力に比例する。infectous profile として、発症日の2日程度前から発症日に向かって急上昇し、発症日後になだらかに減少するような関数(lognormal)を使うと、データをうまく再現できる。発症日よりも前に起きた感染は44%であった。
最後に2つの議論がある。
1.現実の感染させる人ー感染を受ける人の対を選ぶのであるが、感染させる人が発症している場合には人的接触を避ける傾向があるので、感染率が人為的に下がっているということで、serial interval distribution の裾野が低く観測される可能性がある。このことが、infectiousness profile としては、裾野を低くするかもしれない。つまり、この profile には発症者の自粛効果が多少入り込んでいるかもしれない。
2.個別の感染対を選択する時に、より最近に接触した記憶が強く残っている為に、短い interval を選択し易いというバイアスが働いたかもしれない。このことは、infectiousness profile を発症からより前に推定することになる。これらの否定的な可能性もあるが、何よりもPCR検査の結果が、発症前の感染力を示している。
発症日後の PCR検査の推移を見ると、縦軸が Ct値になっている。これは RNA倍増のサイクル数の事で、これが 1大きいということは、サンプル中の初期 RNA量が半分ということである。トレンドだけを見ればかなり早くウィルス量が下がるが、そもそも測定値のバラつきが大きいので何とも言えない。
9月9日
Hao-Yuan Cheng et al.,
Contact Tracing Assessment of COVID-19 Transmission Dynamics in Taiwan and Risk at Different Exposure Periods Before and After Symptom Onset,
JAMA Intern Med. doi:10.1001/jamainternmed.2020.2020, Published online May 1, 2020.
He の論文に対応した台湾でのデータである。スマートな統計理論は使っていないので見やすくはないが、内容的には同等である。
解析対象は 100名の感染者である。感染者と非感染者の間の2761回の接触があった。接触の定義は与えられているが、詳しくは説明しない。この中で、22回だけ2次感染者が生じた。接触回数に対する感染数(これはWHOの定義では発症した場合を数える)の比をattack rate と呼ぶ。つまり感染リスクである。
1次感染者の発症から5日までの間にあった接触回数は1818回で、その内で2次感染者の発症は 1% であった。
それに対して、発症から6日以降の接触回数は852回で、その内で2次感染者の発症は 0.4% であった。
発症前の一次感染者との接触回数は299回で、その内で2次感染者の発症は0.7% であった。(未発症感染者を加えると 0.8%)
接触は家族間接触と施設その他での接触に分類されていて、前者が圧倒的に高い attack rate であった。
発症日前後での感染能力が高いために、COVID-19 では発症者のみを対象にした隔離はあまり有効でないと思われる。
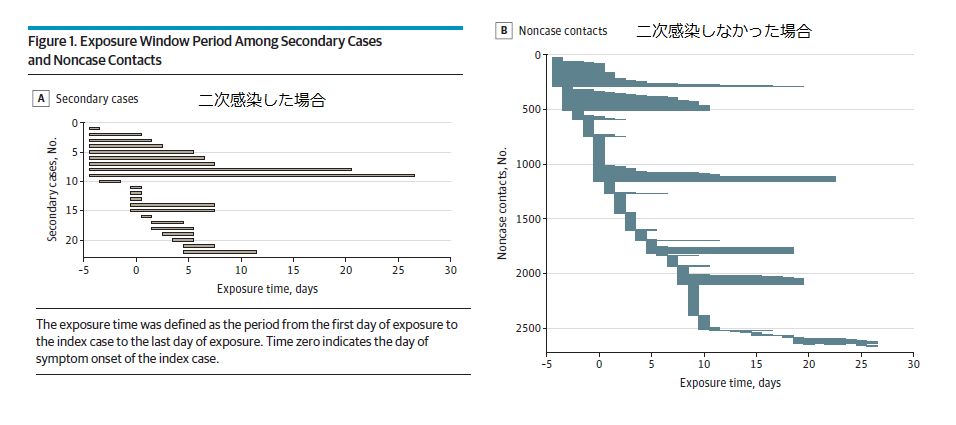
図1A は感染が起きた場合の接触日の分布 図1B は感染が起きなかった場合の接触日の分布
一次感染者の発症日を 0 としている。感染した接触の最初は全て5日までであるが、感染しなかった接触では68%のみが5日までである。6日以降の接触での感染は見られない。(接触の最初の日だけを問題にしているようであるが。。。)
西浦等が報告している serial interval は4〜5日であるが、これが短い原因は、発症日前後の感染能力が一番高いためである。
Nishiura et al., Serial interval of novel coronavirus (COVID-19) infections, Int J Infect Dis. 2020;93:284-286. doi:10.1016/j.
ijid.2020.02.060
PCR検査でのウィルス量の発症日からの推移をいくつかの報告で調べると、発症日から1週間経過するとみられなくなっていることも、本報告と矛盾しない。
一次感染者の発症日前にしか接触しなかった場合が感染していることも明らかになった。
表2には細かい接触日時の分類と接触場所について、感染リスクがまとめてある。香港でのように発症から4−5日経過してから隔離することはあまり効果がなかったであろう。接触履歴を知ることで、発症前での検査に結び付けることが最も効果的である。
9月10日
Jared Bullard et al., Predicting Infectious Severe Acute Respiratory Syndrome Coronavirus 2 From Diagnostic Samples,
Clinical Infectious Diseases® 2020;XX(XX):1–4, published online May 22, 2020. DOI: 10.1093/cid/ciaa638
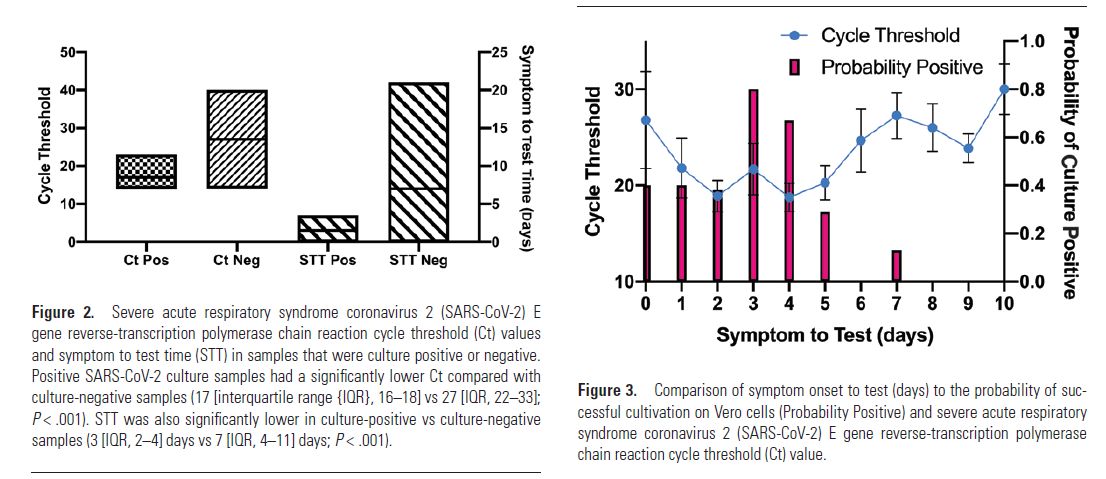
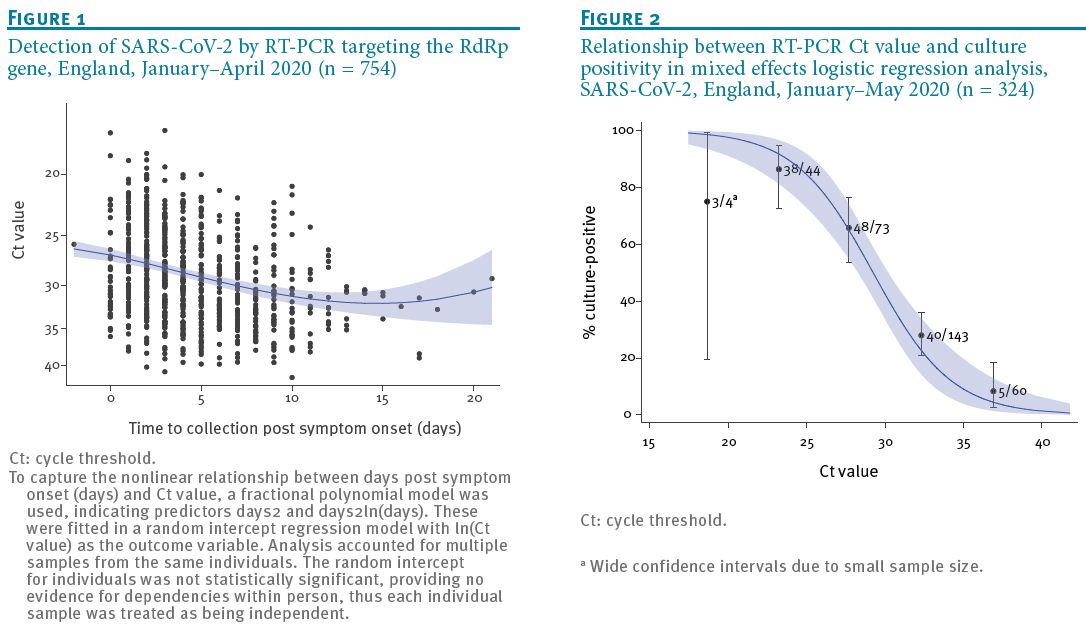
PCR検査は患者からのサンプル中のRNAを検出するのであるが、ウィルスが死んでいても(殻を無くしていても)見つかるから、必ずしも感染力を意味しないことはよく知られている。ここでは、90のサンプルを試験管内で培養して、増殖するかどうか、を調べている(positive-negative)。性別、年齢には依存性が無く、RNA濃度(Ct値:1つ上がると半分である)、発症からテストまでの経過時間(STT)に大きく依存することが判った。図2を見ると良く判る。(この人達の統計処理は初歩的である。)
感染力のある場合の Ct 値での分布を見ると、その平均は 17 で四分位範囲は16-18。(この範囲に半分が入る。)(図の箱には100%が入る。つまり24以上では感染力無し。)
感染力の無い場合の Ct 値分布は 平均が 27(四分位範囲22-33)
同様に、STT の場合、感染力のある場合、STT平均値は3日(四分位範囲 2-4 日)(8日以上では感染力無し。)
感染力の無い場合、STT平均値は7日(四分位範囲 4-11 日)
He 等の結果を支持する結果であり、この場合は患者から採取したRNAの感染性を直接見ているので、発症者の自粛効果は入っていないことが重要である。
隔離解放タイミングであるが、発症から2週間経過した場合には、PCR検査陰性を退院の条件にすることには意味が無いことが言える。
9月11日
Roman Woelfel et al.,
Virological assessment of hospitalized patients with COVID-2019, Nature 581, 465-469(2020)
2020年1月 ドイツの Munich で起きた感染爆発による入院患者の詳細な調査である。著者の関心は、SARSとの比較において、このウイルスの増殖がどの組織で起きるか、ということである。調査対象は比較的少数(10数名?)の中軽症者である。咽頭粘液(throat swab)、唾液(sputum)、便(stool)からのRNA検出と培養によるウィルス検出(isolation)、血液検査による抗体検出。少数の為統計処理は殆どなされていない。
図1を見ると殆どの結論が判る。
a. は発症日起点で日ごと採取したサンプル数である。b. は喉と鼻咽頭粘液に含まれていたRNAの濃度である。c. は唾液と便に含まれていたRNAの濃度である。いずれもバラつきは大きいが徐々に減少している。d. は採取した液からの培養によるウイルス活性の有無である。 生きたウイルスがあったのは、発症日から8日目までである。e. は同じであるが、横軸がRNA濃度になっている。 生きたウィルスがあったのは、log10(RNA per mL)>6 である。f. は d. を活性比率としてプロットしている。 活性のピークは発症日から4日目よりは前にあると思われる。g. は e. を活性化比率としてプロットしている。h. は subgenomic RNA の比率を 各サンプル種別にプロットしている。 subgenomic RNA はウィルスが細胞内で増殖するときに副次的に生じる m-RNA であり、新しく生まれたウィルス中には含まれないから、 これが見つかるということは、その組織中でウィルスが増殖している、 という証拠になる。 唾液中にはかなり長期に亘って見つかるから、口の中で増殖が続いている。 咽頭中では発症から5日目までにしか見つかっていないから、 それ以降、咽頭では増殖していない。 便中にはわずかながら見つかっているから、腸内での増殖はあるが少ない。
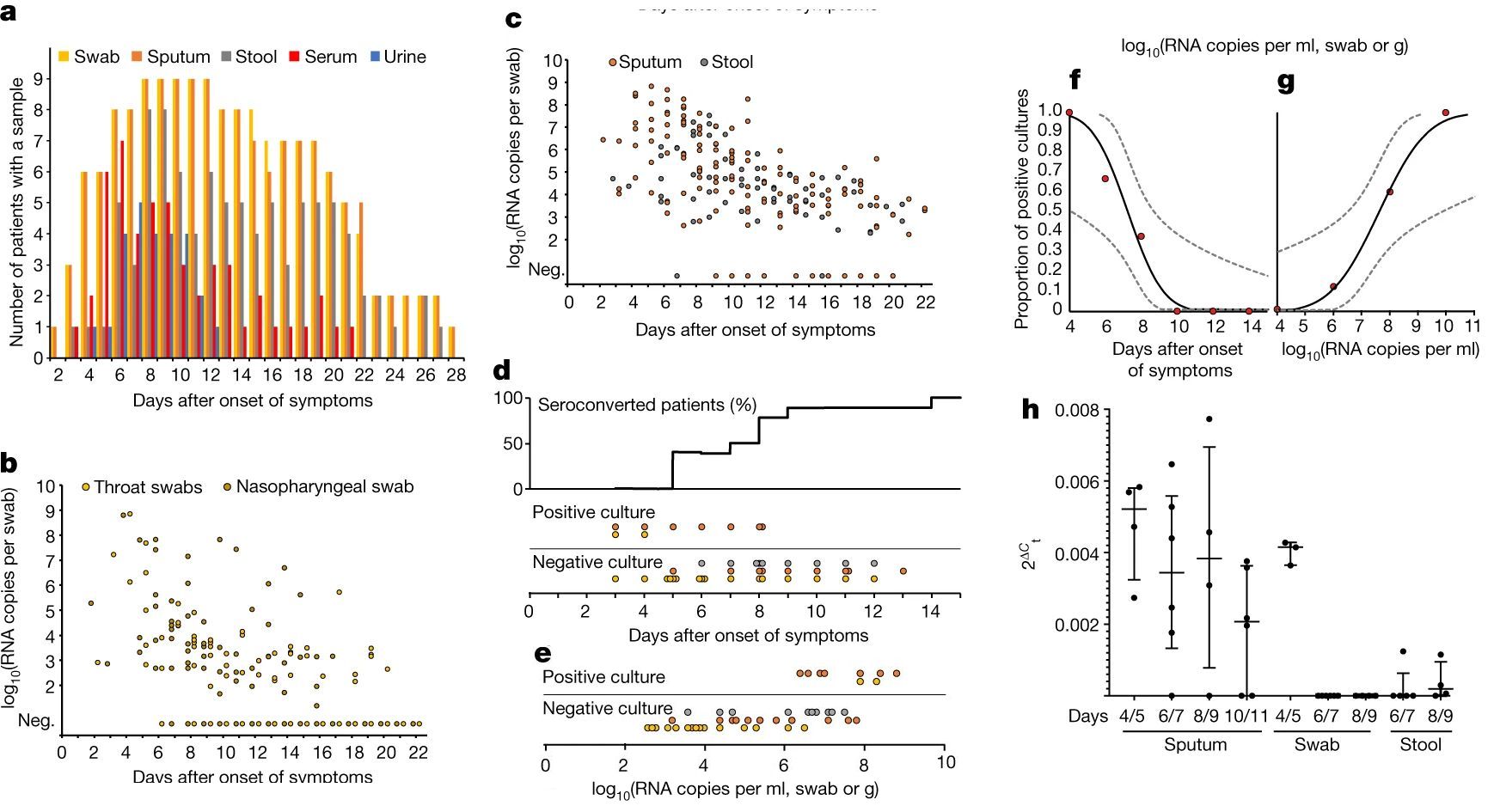
表1には一人の患者で見つかったウィルスの変異データがある。咽頭では変異が見つかったのだが、唾液中には見つかっていない。つまり、増殖は各器官で独立に起きており、肺で増殖したものが単に排出されているわけではない。図2には個別患者のウィルス排出量が発症日からの経過時間でプロットしてある。図1b.c. を個人別に分けたグラフである。また矢印で抗体の出来た日を示してある。9例中6例において、既に症状もなく完全に治癒しているにもかかわらず、3週間を経過してもRNAが検出されている(PCR陽性)。抗体生成は、半分の患者で7日目までに、全ての患者で14日目までに起きている。7日目以降には生きたウィルスは排出されていない。最後に重要な事であるが、この分析には重症者が含まれていない。重症者はもっと複雑な挙動を示すと思われる。
9月14日
感染力の経時変化についての論文は他にも多数見つかる。感染対策には最も重要な情報だからである。発表順。
9月15日
Byrne et al., Inferred duration of infectious period of SARS-1 CoV-2: rapid scoping review and analysis of available evidence for asymptomatic and symptomatic COVID-19 cases,
medRxiv preprint doi: https://doi.org/10.1101/2020.04.25.20079889
これは、感染可能期間のみについての研究の Review である。感染力については別途。
研究には3つの種類がある。
つまり、患者から採取した検体のウィルス活性の研究、
一次感染者と二次感染者の組を多く集めて統計的に解釈した研究、
感染モデルを使ったシミュレーションによる研究。
T2:不顕性感染者についての感染可能期間
T3:発症前での感染可能期間
T5:発症後から治癒(PCR陰性が2回続く)までの期間
T2とT5では回復の意味が異なる点に注意。観測可能ということらしい。
あまりまとまりが無いので、詳しくは読まない。
Casey et al., Pre-symptomatic transmission of SARS-CoV-2 infection: a secondary analysis using published data,
medRxiv preprint doi: https://doi.org/10.1101/2020.05.08.20094870
同じく Byrne のグループによる文献調査で、発症前感染可能期間の Review である。
Mc-Evoy et al., The relative infectiousness of asymptomatic versus symptomatic infected persons with COVID-19 – A review of data available until 8th April 2020. 2020.
同じく Byrne のグループ。これは削除されていて、8月に再度投稿されている。中身は4月1日まで。
Mc-Evoy et al., The relative infectiousness of asymptomatic SARS-CoV-2 infected persons compared with symptomatic individuals: A rapid scoping review.
August 2020
https://www.medrxiv.org/content/10.1101/2020.07.30.20165084v1.full.pdf
不顕性感染者の感染能力についてはあまり研究が無いので、不確かである。著者としては発症感染者に比べて0.4〜0.7程度ではないか、と考えているが、少数の実例からの推定なので、0 あるいは 1 の可能性すら残る。表1に数少ない個別の論文の結果がまとめてある。今後の研究が期待される。
9月16日
Chun et al., Transmission onset distribution of COVID-19,
International Journal of Infectious Diseases 99 (2020) 403–407, 21 July 2020
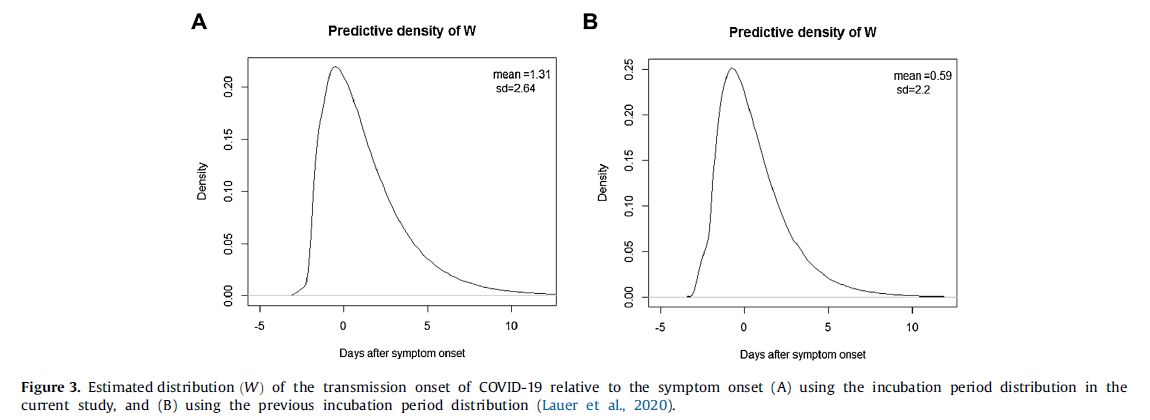
72例の一次感染者(infector)二次感染者(infectee)対と潜伏期間分布データを組み合わせて、発症日基準での感染日の分布を求めた。(He と似ている)。感染日のメディアンは発症日から1.31日後で、標準偏差は2.64日。そのピークは発症日の0.72日前。発症前感染の比率は37%。潜伏期間のメディアンは2.87日、serial interval のメディアンは3.56日。
S:発症日、I:感染日、P:感染可能になった日、(CL〜CR):接触日の範囲
W=P−S
j をinfector、i をinfectee として、データは (Sj,CL,CR,Si)と記述される。
CL≦I≦CR。Pj=min Ii。W の分布は Pj−Sj を集めれば推定できる。
データの状況は次の3種類
(i) I= CL=CR;(ii) CL≦I≦CR; (iii) CL=−∞、CR=S (家庭内感染)、I が判らない。
Wについての分布関数 f(W) を求めたい。
( f(I−S) ではない! He et al. とは違うが、あまり多くの人に感染させている訳でもないから、実際上は殆ど同じだろうと思う。)Bayesian methods を使うということである。parametric model の式
W 〜 f(・|θ) ⇒ W+μ 〜 πg(・|φ)+(1−π)U(−D,0)
分布関数の形を決めるパラメータが θ と総称される。ガンマ分布として、そのパラメータ2つを φ と総称して、その分布をずらす値として μ もパラメータである。さらに π を使う。これは、都合の悪い分布値、つまり分布の裾を切り落とす為のものらしい。(He et al. では切り落とされていないのでこの辺が改良点。ベイズ法だと簡単らしい。)W〜f(・|θ) というのは、W の分布が f(W|θ) である、という意味のようである。普通は分布パラメータ θ を持つ分布に W が従う、という意味であるが、これを θ という前提条件の元での W の分布と考える。逆に、観測された W からパラメータ θ を決めるのであるが、普通の統計では θ を変えてみて、f(W|θ) が最大値を採る(尤度最大)ように決める。ベイズ推定では、θ にあらかじめの予測分布を与えておいて、その主観分布を実際の W によって修正する、というやり方をする。データが少ない場合には、その方が信頼できる。(この「信頼できる」というのは「主観的に」である。)要するに、限られたデータから分布を推定するという行為は何らかの主観無しには実現できない、という「現実に即した」やり方である。もっとも、そもそも分布関数の形を決めるということ自身が主観的である。MCMC(マルコフチェインモンテカルロ)というシミュレーション的な計算方法でやる(分子系のシミュレーションではよく使われる。一回毎に乱数で2者択一を行って、その繰り返しで最終的に望む分布が実現するような試行を繰り返すやり方)。詳しくは別ファイルで説明してある(Rのコードが書いてある)ということだが、wordファイルになっていて、word2003 に変換しようとすると PC がフリーズしてしまうので判らないが、まあ技術的な問題に過ぎないだろう。実測の W は上記 (i) でのみ確定する。(ii) (iii) については、Si−I の分布、つまり実測の潜伏期間分布から乱数発生で作り出す。感染性発生分布は図3である。He とあまり違わない。分布の左端が負になっていて、そこが −μ である。
9月19日
Jefferson et al., Viral cultures for COVID-19 infectivity assessment – a systematic 2 review (Update 3), medRxiv preprint doi: https://doi.org/10.1101/2020.08.04.20167932
定期的に更新されている PCR検査における RNA量とウィルス活性 の Review である。
RNAの排出(viral shedding)、ウィルス生存(live viral culture) と Ct サイクルの関係、さらには、発症からの日にちの関係である。
注目される、発症からのウィルス生存日数については、3つの報告(Woelfel、Bullard、Singanayagam)があり、前2者では8日以上、後1者では11日以上では生きたウィルスが無い、というのが共通している。RNA排出については長い。14日から35日というのもある。
生きたウィルスと Ct との関係については、その最高値として 24とか34がある。Ct が一つ大きくなる度に、ウィルス生存の Odds 比が 0.64、0.67 づつ下がる。
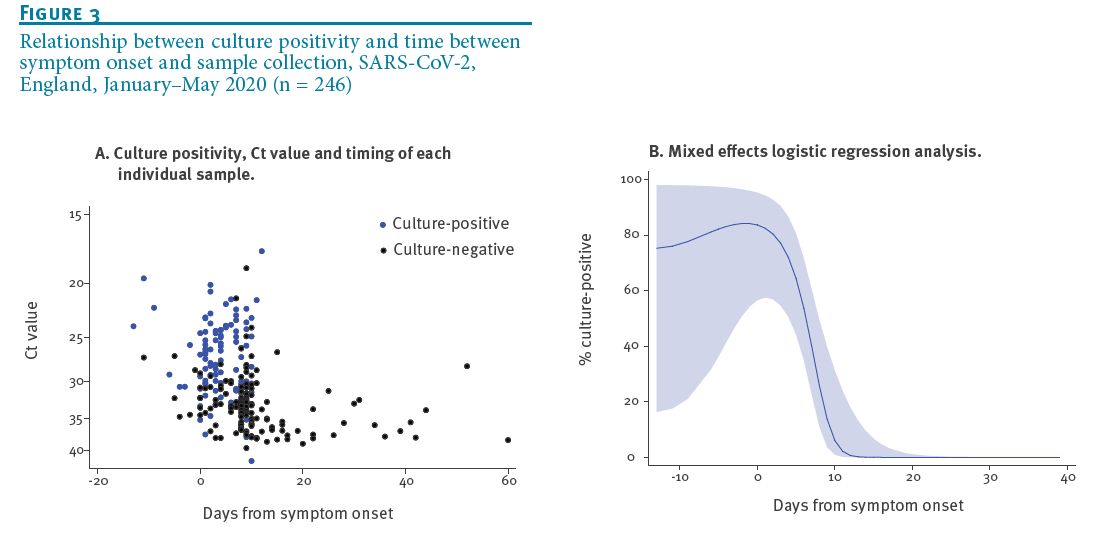
Singanayagam et al., Duration of infectiousness and correlation with RT-PCR cycle threshold values in cases of COVID-19, England, January to May 2020, Euro Surveill. 2020;25(32):pii=2001483. 13 Aug 2020
https://doi.org/10.2807/1560-7917.ES.2020.25.32.2001483
ウィルス活性については一番新しい報告である。図3を見ると結論が良く判る。発症から9,10日目あたりで急激に活性が無くなる。その後だらだらとRNAが排出される。Ct については 図1、図2が判りやすい。活性は Ct=28辺りを境に急激に下がる。Ct そのものは 大きくばらついているが傾向として 8日目あたりで30を超える。
9月20日
Ferretti et al., The timing of COVID-19 transmission, medRxiv preprint doi: https://doi.org/10.1101/2020.09.04.20188516
(このpreprint では、図の再利用が許可されていないので、ここでは掲載しない。原論文を参照のこと。。。)
一次感染者が二次感染者に感染させるタイミングについての最新の研究である。感染タイミングは一次感染者の感染日よりもその発症日に関係している。発症日の前後2〜3日の間に75%以上の感染が生じている。発症前感染の比率は41%である。ただし、注意すべき点の一つは、感染から発症までの時間(潜伏期間)の広い分布であって、潜伏期間が長い場合には、発症の2〜3日前よりも更に前で二次感染が起きている場合もある。また、二つ目として、発症後の二次感染については、発症による行動の自粛が影響していて、感染力が弱っていくという事だけでは無いので、注意が必要である。いずれにせよ、発症前の検出が重要であるから、何らかの検出方法の開発が期待される。
不顕性感染者からの感染については、発症する感染者に比べて感染力が低いという間接的な証拠がある、として文献を挙げているので、当たってみる必要があるだろう。
コンタクトアプリについても触れていて、発見された感染者から個別に聞き取るよりは効率的に未発症感染者を見つけられるが、人口あたりどれ位アプリが普及しているか、どれくらいコミュニティの中で共有されているか、感染可能性が表示されてからどれくらい早く検査ができるか、に依存する、として文献が挙げられている。
感染者の早期発見手段の効率的な運用の為にも、感染リスクの期間を限定することが有用である。だからこの研究に意味がある。
4つの期間データがあり、4つの感染日データと以下の関係がある。(これは図1 になっている。)
the incubation period(潜伏期間)
the serial interval,
the generation time(latent period)
the time from onset of symptoms to transmission (TOST)(正と負がある)
一次感染者の感染日, 一次感染者の発症日, 二次感染日、二次感染者の発症日
↓ 潜伏期間 ↓ TOST ↓ 潜伏期間 ↓
↓ serial interval ↓
↓ generation time ↓
・serial interval のデータは 5つの文献から得た。
平均5.1日、メディアンで4日、標準偏差で3.8日。対数正規分布でよく合う。付録図1。
・incubation time
7つの文献から、平均5.7日、標準偏差3.5日。
・generation time
最尤法で最適な分布関数形を探した。注意したのは、感染日(t=0)近くでほぼゼロになる(何故ならばウィルスの排出がほぼ無い)ことである。赤池基準で最もよかったのはWeibull分布である。図1A:平均で5.5日、標準偏差で1.8日。Gompertz, a log-logistic and a gamma distributionもこれに近かった。これらは付録図2。 generation time と serial interval とは、図示するとずれているだけのように見え、実際、近似的には同じとされる場合もあるが、一般的には異なる。
・TOST
同じく最尤法で、最適だったのは、Student’s t distribution で、平均-0.07日、標準偏差2.8日、図1B。skewed logistic 分布もそれに次いで良かった。
・generation time と TOST のどちらに意味があるのか?
赤池基準で計算すると明らかに TOST に意味がある。つまり、二次感染は一次感染者の感染日よりも、発症日に強く依存している。 つまり、潜伏期間の分布はかなりばらついている。これに対して、発症日はウイルスの活動時期をかなり正確に反映している。
・感染日は影響が無いのか?
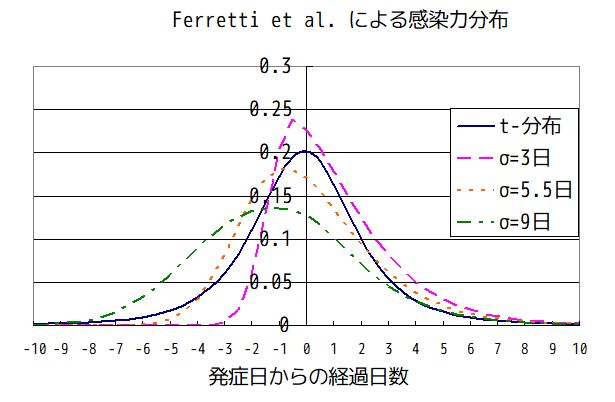
潜伏期間とserial interval の間には弱い負の相関が見られた。つまり潜伏期間が長い場合には二次感染者の発症日がやや早まる。これは、一次感染者の発症日だけで感染が決まっているとすると起こりえないことである。一次感染者の感染日から発症日にかけて少しづつ感染確率が上がっていることを示している。つまり TOST分布の負側は潜伏期間が長くなると少し引き延ばされて、感染力が早めに現れる。 図3B。下図は論文中の計算式に従ってエクセルで計算した感染力分布である。t-分布は全てのデータを同等に扱った場合の分布(図1B)で、他は潜伏期間を指定した分布である。σ は潜伏期間を意味する。この場合、skewed ロジスティック分布を使い、発症日より前の分布だけを潜伏期間に比例して時間軸を引き延ばす、という方法で最適化している。分布は全て積分値が 1 となるように規格化してある。潜伏期間分布について平均すると、ほぼ 全体の分布に近くなるが、想定した関数形が異なるので一致はしない。感染を受けてから徐々に感染力が上がっていき、飽和したあたりに発症する。その期間の長さについては広い分布があり、それはウイルス侵入量や自然免疫に左右されるのであろう。場合によっては発症しないで終わる(不顕性)。
・presymptomatic transmission
単純に TOSF から計算すると41%。元のデータに戻って、潜伏期間を考慮したモデルでベイズ流に推定しても41%。(このベイズ流の意味は良く判らない。)発症当日と翌日の感染は1/3位である。ベイズ流で推定すると35%。翌々日以降の感染は22%(ベイズ流で24%)である。しかし、この辺になると、患者の接触回避行動が影響していると思われる。少しでも疑わしい症状がある人が接触を避けるようにすることで、かなりの感染予防効果がある。感染可能性が生じている時に少しでも早く検査をすることが重要である。(なお、この計算の時には、感染力分布を一日毎に棒グラフにして、0 日目は presymptomatic transmission には入れていない。これは良いやり方だと思う。)
Tao et al., Determination of risk factors for predicting the onset of symptoms in asymptomatic COVID-19 infected patients,
Int. J. Med. Sci. 2020(16 Aug), Vol. 17, pp.2187
無症状感染者としてPCR検査で見つかった患者の分析である。
武漢での初期のデータで、平均年齢は若い(33歳)。70名中隔離中に発症したのは22名で、未発症は48名。妊娠中の1人を除いて、胸部CTで異常が認められたのが35名。重症者は無し。PCR検査陽性確定から陰性になるまでの平均日数は13.2±6.84日。(つまり、発症日からだともっと短い。)統計解析で認められた発症相関は喫煙歴、心臓疾患であった。また、白血球減少、リンパ球減少が前触れとしてあることが多い。PCR検査陽性判明から発症までの平均日数は4.41日±3.55日で、1日ー14日の分布がある。下図参照(横軸は陽性確定日からの日付)。
若い人の感染は無症状のまま推移する場合(不顕性感染)の方が多いということが判る。67%。CTでも50%。これは全感染者に対する比率ではなくて、あくまでも検査陽性時無症状陽性者に対する比率であることに注意。

Lee et al., Clinical Course of Asymptomatic and Mildly Symptomatic Patients with Coronavirus Disease Admitted to Community Treadment Centers, South Korea,
Centers for Disease Controle and Prevention, Vol. 26(10), October 2020
これは Preprint で、韓国の政府機関の機関誌の10月号に公表予定らしい。
未発症・軽症患者数は632名。75名が最初から発症、186名が隔離中に発症、371名が発症せず(不顕性感染者)。19名が病院に搬送されたが、573名はそのままPCRテスト陰性となって治癒。陽性の期間の平均は20.1日、標準偏差は7.7日。発症者の方が未発症者よりも長い傾向あり。軽症者の場合、発症からの陽性期間(virological remission period)平均は11.7日、標準偏差は8.2日。(この abstract が正しいのだろうが、以下内部では この2倍の日数が記されていて定義が良く判らない。)
2月後半に見られた宗教団体での感染爆発を抑える為に、団体の全員が隔離されて検査された。10,459名中4,259名が陽性であった。隔離と治療体制が追い付かなかったので、3月に入って、community treatment center を作り、無症状+軽症者を隔離した。今まで、入院に至った患者についてしか報告されてこなかったので、今回は CTC に入った軽症者の経過を報告する。
分類法
無症状:50歳以下、基礎疾患無し、非喫煙、37.5℃以下、解熱剤無し。
軽症:50歳以下、基礎疾患有り、38℃以下、解熱剤。
重症:意識有り、呼吸困難 あるいは 解熱剤を飲んでも 38℃以上。
重篤:意識障害有り。
632名。平均年齢は40.6歳。112名に基礎疾患有り。患者はCTCに入る前、検査陽性から平均で7.8日自宅で待機した。検査時に発症は31名、その時に無症状でCTCに入るまでに発症したものは44名。残りはCTCに入るときには無症状であったが、その後発症したものは186名で、最後まで発症しなかった者は371名。発症分類は表1。19名は重症化して病院送り(表2)。残り578名には粘液採取と検査を繰り返した。陰性までの日数は平均20.1日(7〜45日)。1週間以内が2名、2週間以内が157名、3週間以内が362名、4週間以内が489名、5週間以内が550名。
症状悪化で病院送りになった者を除いて、67名がCTCに入る時から発症、175名は陰性で後に発症。371名は無症状のまま。陰性化の時間は、それぞれ、平均19.6日(7〜43)、23.1日(8〜45)、19.1日(7〜45)。この日数は陽性確定日を起点にしているものと思われる 。潜伏期間が長いのでこういうことになる。
613名中242名が発症したが、その発症日から陰性化までの時間は、平均で11.7日(2〜41)。1週間以内が90名、2週間以内が149名、3週間以内が188名、4週間以内が207名、5週間以内あるいはそれ以上が219名。
ほぼ全数検査であるので、重要なデータである。平均年齢40歳、軽症者、ということであるが、最後まで無症状のまま(不顕性感染者)の比率が 59% というのは、こういう研究でしか得られない。ただ、この比率は全陽性者に対する比率ではなく、CTCに入った陽性者に対する比率である。また、どの程度の感染能力があるのかは判らない。実験する訳にも行かないだろうし。。。
全体を眺めてみると、PCR検査陽性確定から、発症までの日数分布が凡そ推定できる。発症者は全部で261名。検査時に発症していたのは31名、その後1週間自宅待機中に発症したのは44名、CTCに入ってから発症したのは186名である。つまり、未発症陽性者が陽性確定から発症するまでにはかなりな時間がかかっていることが判る。これは Tao 等の論文(4.41日)より長いようである。Ferretti の論文によれば、潜伏期間が長い場合には、感染力分布も発症日よりかなり早めに立ち上がることが知られているので、そういう状況が起きていると考えられる。無症状感染者については、他にも韓国発の非常に多くの論文があるので、調べてみることにする 。









