primer 2 (20pmol/
dNTP(1-2mMのStock solution)1-2
PCR buffer (Mg Cl2 15mM, gelatin freeのもの), 10l
template plasmid 数ng(10ng/
条件に応じて場合によっては25mM Mg Cl2を1-4
AmpriTaq 0.5
primer 1 (20pmol/l), 2
l
primer 2 (20pmol/l), 2
l
dNTP(1-2mMのStock solution)1-2l
PCR buffer (Mg Cl2 15mM, gelatin freeのもの), 10l
template plasmid 数ng(10ng/l程度の濃さに薄めておくのがよい。)
条件に応じて場合によっては25mM Mg Cl2を1-4l加える。
AmpriTaq 0.5l
合計100lとする。
*Reactionを多数行うときはこれらを必要に応じてpre-mixする方がAmpriTaqの節約、また実験時間の短縮にもなる。いずれにせよ最初に条件を書き出してから進めること。
PCR program(代表的なもの)
94℃ 45secメモ:
55℃ 30sec
72℃ 30sec
25 cycles後
72℃ 7min
4℃ soak
PCR reactionは全体を20lのボリュームで行ってもほとんど問題ない。このばあい、AmpriTaqは20/100X0.5=0.1_lでよい。
primer 1 (20pmol/l), 1-2
l
primer 2 (20pmol/l), 1-2
l
dNTP(1-2mMのStock solution)1-2l
PCR buffer (Mg Cl2 15mM, gelatin freeのもの), 10l
template 1st PCR product 5l(gel purified:gel isolationの項を参照)
条件に応じて場合によっては25mM Mg Cl2を1-4_l加える。
AmpriTaq 0.5l
PCR program(代表的なもの)
94℃ 45sec
55℃ 30sec
72℃ 30sec
25 cycles後
72℃ 7min
4℃ soak
QIAquick PCR purification kitのマニュアルを参照してください。
簡単にいうと:
1. PCR product (100l) + 500
l PB bufferを混ぜて紫色のカラムに入れる.
2. 紫色のカラムを 2mlの付属の白いtubeに載せ、短く強遠心する。
3. 白いtubeの中身を捨てPE bufferを700lカラムに入れ、短く強遠心する。
4. 白いtubeの中身を捨てカラムを空のまま短く強遠心して乾かす。
5. カラムに50lのTEを入れてエッペンドルフチューブに強遠心で溶出する。
ここではいくつかの例に沿って解説します。
Vector
pGEM系、pBluescript系のベクターは
use 5g DNA
pSV系のベクターに関しては
use 10-15g DNA
Insert
1 から 2 PCR reactionの全量使用
Digestion
の手順NgoMI & BbS I
use NEbuffer 2, NgoMI digest, then BbS I digest
BbS I & Bgl II
use NEbuffer 2, BbS I digest, then Bgl II digest
Bgl II & Bpu1102 I digest
use NEbuffer 2, Bpu1102 I digest, then Bgl II digest
Bpu1102 I & Kpn I digest
use NEbuffer 2+1l NEB BSA (also in yellow buffer box), Bpu1102 digest, then Kpn I digest
ポイント:
1. トータルのVolumeは60l前後が望ましい。
2. Kpn Iなどはstar activityが強いため、37℃, 30-40分とし、Double digestの場合は必ず後から加えて切断する。これで十分切れる。
3. NgoMIは切れにくいことがあるため注意する。
ここではいくつかのポイントのみ挙げる。
1. 泳動するフラグメントの大きさに合わせてゲル濃度を調整するとよい。
10Kb以上:0.7%
1.5-10Kb : 0.7-1%
1Kb以下:1.5%
100bp以下:必要に応じて3%Nusieve, 1%agarose
2. 特にゲル切り出しなど、またDNA量が少なそうなときはゲルが必要以上に厚くならないようにする。
3. 制限酵素切断後のサンプルにはグリセロールが少なからず入っていてきれいに泳働できないことがある。この場合は前もってサンプルを70℃以上で5分程度heat upしておくと多少きれいに流れる。
QIAEX IIのprotocolに従ってください。
具体的には、
1. 50℃の恒温層を準備
2. アガロースゲルから適宜切り出し(多すぎないように)、エッペンドルフチューブに入れ、QX1 bufferをトータル1-1.2mlになるように入れる。
3. QIAEX gelを10-15l入れて恒温層のなかで転倒混和しながら10分インキュベートする。
4. ゲルが完全に溶けたことを目視で確認し、強遠心する。
5. ペレットをQX1で1-2回洗う。
(洗浄はボルテックスを用いてもよいが10Kb以上の大きなフラグメントの時はP-1000ピペットで注意深くペレットを崩した方がよい。)
6. ペレットをQEで1-2回洗う。
7. 最後の遠心後、わずかに残ったQEを注意深く吸い取ってから15分程度乾かす。
8. 20_lのTEを入れ、室温もしくは50℃で5分インキュベートして溶出後強遠心。実際には16-18lになる。
9. 2nd PCR用などすぐに用いない場合は-20℃で保存。
9. 特にInsertはすぐにLigationに用いるのが望ましいが、できなければ-20℃で保存。
Vector
pGEM系のベクターに関しては
use less than 1g DNA
具体的には
5_gのQIAEX IIによる Vector preparation ~20lの内、0.5
l程度を使う。
pSV系のベクターに関しては
use 1-2g DNA
具体的には
10gのQIAEX IIによる Vector preparation, ~20
lの内、1-2
lを使う。
Insert
QIAEX IIの溶出液16-20lを全量使う。
1. NEB or BoheringerのT4 DNA Ligaseを使用する方法
Mixtureの組成
Insert (16l)
NEB ligation buffer (2l, 青いboxにあるものを小分けして使う。)
NEB T4 DNA ligase 1l
反応は16度または室温でovernight
2. TAKARA ligation kitを使う方法
TAKARA ligation mitture I を等量のInsert (16-20l)と混ぜる
16℃ 30min
注意:
1. Ligation mixtureは4℃で保存できる。
2. できるだけInsertを入れない(つまりH2OやTEで置き換えた)
3. negative controlのligation mixtureを1本作って一緒にTransformationさせること。Backgroundのコロニー数がわかる。(次次項参照)
1. put freezed competent DH5 cell (50l each)on ice for 5-10min to defreeze
2. put 2-5l ligation mixture into cells
3. place on ice for 30 min
4. prepare 42℃ water bath, prewarm LB medium (necessary volume), and prewarm LB plates containing antibiotics (for ampicilin, 100g/ml)
5. heat shock for 60sec
6. immediately place on ice for 2min
7. put prewarmed 0.5ml LB, transfer to 15ml tube, then shake for 1 hour
8. centrifuge breifly (1500rpm, 3min)
9. aspirate ~0.5ml LB and resuspend pellet
10. spread bacteria onto LB plate containing antibiotics
11. 16-24 after, pick up colonies and put into 5ml LB medium containig antibiotics (for ampicilin, 100g/ml)
12. shake overnight to get confluent bacterial solution
1.stock大腸菌を抗生物質(ー)プレートにstreak
2.コロニー1個を5mlLBで一晩culture
3.菌液を1ml/100mlに希釈してOD550を15分ごとにチェックしながら37℃約2時間shake
4.OD550 = 0.5前後のところでフラスコを取り出しChill on ice, 5min
5.以下の処理は厳重に冷やしながら行う
6.50ml Falcon tube(あらかじめ冷やしておく)で約3000rpm, 5min
7.ペレットを40ml Tfb I (4℃チャンバー、あらかじめon iceで冷やしておく)に優しくsuspend
8.Chill on ice, 5min
9.50ml Falcon tubeで約3000rpm, 5min
10.ペレットを4ml Tfb II (4℃チャンバー、あらかじめon iceで冷やしておく)に優しくsuspend
11.Chill on ice, 15min
12.1.5ml エッペンドルフチューブ(あらかじめon iceで冷やしておく)に50オ;lずつ分注
13.-80℃に保存(レブコのなるだけ深いところに)Blue-White Screen用のPlateの準備
プレート1枚あたり30ml LB agar (12-15g/L agar)
まとめてオートクレーブして固まらないうちに使用する。熱すぎるのはよくない
Ampicilin, 100g/ml (25mg/mlのものを250倍希釈)
IPTG stock ~20mg/ml, ~30l
X-gal stock: 20-100mg/ml, ~30l
冷え固まったら、パラフィルム、遮光して4℃保存
1. negative controlと比較してあまり差がない場合は多めにコロニーを拾った方がよい。(4個以上)
2. Yellow tipを上手に使って寒天と一緒にコロニーを吸い出し、100g/mlのAmpicilin(Ampicilinで選択する場合)を含む5mlのLB培地にresuspendする。
3. bacteria shakerで16時間以上37℃で培養する。
I. QIA-prep PCRを用いる方法
1. 1-1.5mlの培養液をエッペンドルフチューブで遠心(3500rpm, 3-5min)
2. Pelletを~200l P1 bufferにresuspend
3. ~200l P2を加えて混和(5分以内で)
4.~ 200l N3を加えて混和(強く振りすぎないように)、強遠心。
5. 上清をカラムにapply、短く強遠心(卓上ミニ遠心器を用いてもよい)
6. (大腸菌の種類によっては;プロトコール参照)PB bufferで洗浄、遠心
7. 750lのPE bufferで洗浄、遠心
8. 遠心によって乾燥
9. 100lのTEを加え、1分ほど待って遠心して溶出
II. Wizard mini-prepを用いる方法
この方法が収量がよく精製度もこの中では一番優れている。
1. 1-1.5mlの培養液をエッペンドルフチューブで遠心(3500rpm, 3-5min)
2. Pelletを200l Cell resuspension solution(赤い容器)にresuspend
3. 200l Cell lysis solution(黄色)を加えて混和(5分以内で)
4. 200l Neutralization solution(青)を加えて混和(強く振りすぎないように)、強遠心。この間にVacuum manifoldにカラムとシリンジをセットしておく。
5. 上清をシリンジ上にapply、吸引を開始。
6. 液が無くなったら2mlのColumn wash solutionをシリンジ上にapplyし吸引を続ける。
7. 液が無くなったらカラムだけはずし、エッペンドルフチューブにセットして短く強遠心して乾かす。
8. 50l TEを入れて1分ほどおいてから強遠心して溶出する。
III. 8M Ammonium acetateを用いる方法
多少時間もかかり精製度、収量ともいまいちだがPCRをかけてスクリーニングするには十分。なんと言っても安価なので大量にやるときは良心の仮借を感じずにすむ。
1. 0.5-1.5mlの培養液をエッペンドルフチューブで遠心(3500rpm, 3-5min)
2. Pelletを60lの200
l TNE (10mM Tris, pH 8.0, 100mM NaCl, 1mM EDTA)にresuspend
3. 60lのPhenol/Chloroform isoamyl alchol (25/24/1)を加えて混和。
4. Sampleを冷却強遠心(約3分)、60l上清をあらかじめエッペンドルフに入れておいた20
lの8M Ammonium acetateに入れ、vortexする。
5. ~160l cold EtOHを入れ on ice15min沈殿させる。
6. 15min 強遠心、ペレットを75% EtOHで 洗って再遠心してスピードバックで5-10min乾かす。
7. 10-20lのTEにresuspendしてPCR templateとする。
コロニーが当たりだったらやることがあります。
1. 当たりコロニーに終濃度7-10%のDMSOまたはglycerolを加え-80に保存。これを使用するときは、
a. 大腸菌氷一かけらを白金ですくいとってAmpicilinなどが入ったプレート上に広げる。
b. 翌日コロニーを一つとってminiprepして使用する。保存大腸菌から直接とってminiprepしてはいけない。
2. 当たりのmini cultureは4℃でしばらく保存できる。
Sequenceをより確かなものにするためM13 vectorにInsertをクローニングすることがある。M13ファージはf-piliを持つ大腸菌(今回はDH5aF'を使用する。)に感染し、Host E-coliを溶菌させることなくHostからcapsidに包まれたファージ粒子を分泌させる。ファージ粒子内には一本鎖DNAが含まれ、これを後に述べる方法で単離することにより、sequence反応に最適な一本鎖templateを得ることができる。M13はプラスミドのような二本鎖DNAとして大腸菌内に存在する(Replicative form, RF)ので、RFに目的DNAをligateすればrecombinant phageが得られる。
作業の流れ
1.あらかじめRFをblunt enzymeであるSma Iで切断、T-vectorとして調整しておく。(T-Vectorの作成 の項参照)
2.PCR productをTA-cloningでLigation、これを以下の方法でtransformして感染大腸菌のプラークを得る。プラークは4℃で保存できる。
3.保存しておいたプラーク(ファージが入っている)を再感染(混ぜてインキュベートする:2.)させて培養上清にファージを発芽させ、これをいったん4℃で保存、大腸菌ペレットからRFを調整して(Mini-prep of each colonyの項参照)Insertをアガロース電気泳動で確認する。
4.4℃で保存しておいた上清からいかに述べる方法でsingle strand DNAを回収する。
5.DNA量をゲルで測定し、Auto-sequence反応を行う。
1. Plating bacteriaの準備:DH5aF' cellをLB plate (without ampicilin)上にstreakしたものを前もって4℃に保存しておき、前日プレートからコロニーを一つとって5mlのLBに入れ、overnightで増やしておく。
2. 当日あらかじめ以下のものを用意する。
42℃ water bath
Top agar (7g/l in LB: 通常のLB-agarをLBで半分に希釈、4の各チューブそれぞれに対し3ml必要なので必要量を42_ water bathにつけておく。)
prewarm LB plates (without ampicilin)、ラベルしておく
3. put freezed competent DH5aF' cell (50_l each)on ice for 5-10min to defreeze
4. 2-5l のligation mixture を4mlチューブに入れ、3をゆっくり吸って静かに加え1ー2度tappingして軽く混和する。
5. place on ice for 30 min
6. 7の直前にplateをincubatorから出し、キムタオルを敷いた上に蓋を上にして並べる。
6. 5をheat shock, 42℃for 60sec
7. immediately place on ice for 2min
8. その後直ちにTop agarに以下のものを混合する。4の各チューブそれぞれに対し3ml必要なので、少し多めに必要なだけ調整する。
X-gal (final 約250g/ml)
IPTG (final 約250g/ml)
Overnight plating bacteria, 200l for each tube
9. 8が固まらない内にすばやく8から約3mlずつ取って7に加え、転倒混和して全量を出しておいたプレートの上に蒔く。プレートを傾けて薄くtop agarを広げる。
10. 固まったら逆さまにしてインキュベーターに入れる。
11.12-16時間後に観察してwhite plaqueを爪楊枝で取って0.5-1mlのLBに浮遊させる。この状態で4℃数カ月保存できる。
- Plating bacteriaの準備:DH5aF' cellをLB plate (without ampicilin)上にstreakしたものを前もって4℃に保存しておき、前日プレートからコロニーを一つとって5mlのLBに入れ、overnightで増やしておく。
- 1. Transformation of DH5aFで用いたcellが余っていればこれを用いてもよい。
- . 2-5mlのLBに上記bacteriaを40倍希釈する。
- . 1-11の保存プラークの1/10を取ってこれに入れ、5-8hrs振盪培養する。
- 1.5mlをエッペンドルフに移して遠心し、上清を別に保存する。
- ペレットをWizard mini-prep法で処理して(Mini-prep of each colonyの項参照)菌体よりRFを抽出してenzyme cut, アガロースゲルで確認する。
B. Single strand DNA
の調整QIA prep spin M13 kitに従う。
PCR用チューブを用いる。
primer: 3.2-3.7 pmol
template DNA: 0.5g(正確に定量する。ここが最大のポイント。多すぎてもいけない。)
プレミックス8l(あらかじめ分注したもの)
dH2Oで合計20lにする。
PCR machine (9600または2400)
96℃, 10sec
50℃, 5sec
60℃, 4min
(25 cycle)
以後4℃、遮光保存
2. Sequence反応産物の精製
A. EtOH沈殿法
上記反応産物20l
3M NaOAc 2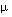
以上をよく混ぜ合わせ95% エタノール 50l を加える。
以上を混ぜ合わせて
室温30min
13000rpm, 20min、室温で遠心する。4℃で遠心するとペレットが溶かしにくくなる。
ペレットにさわらないように注意深く上清を捨ててから75%エタノールでペレットを洗浄
13000rpm, 10min遠心
ペレットにさわらないように注意深く上清を捨てSpeedVacで10min dry-up
25のTemplate suppresion reagent (TSR) にpipettingでよく溶かしてDNAを回収する。
Sequencerにapplyするまで4℃または-20℃で保存する。
3. Automatic sequencerにsampleをapplyする。
3-1. Sampleを95℃、3minHeat-upする
3-2. 右側のドアを開けsampleをapplyする準備をする。
現在使用中でないことを確認する。
dH2Oチューブが空になっていたら継ぎ足す。
ゲルが無くなっていないかどうかチェックする。
Sampleを専用チューブに移し蓋をしてカセットにセットする。
ドアを閉めて一度Macintoshを再起動する。
ABI Prism 310 Collectionをオープンする
FileメニューからNewを選択
Create New sequence Smpl Sheet 48 Tubsを選択
Sample sheetがでたらsample名を入力する。
FileメニューからNewを選択
Create Sequence Injection Listを選択
File menu barからsequence injection listを選択する。
window menu barからmanual controlを選択する。
Quiagen Maxi Kitが最適である。
1. 前日125-250mlのLB broth (100g/ml Ampicilin)にminiprep大腸菌を100-500倍希釈程度に入れ、16-24hour 37℃で一晩shakeする。
2. 5研遠心機を2時間半ぐらい確保する。大腸菌を250ml遠心管に入れ、5研遠心機で4000rpm程度で15min, 4℃遠心
3. ペレットを電動pipetterを用いて10mlP1 bufferで溶かし、50ml専用遠心管に移す。
4. 10ml P2 bufferを加え、できるだけ完全に溶菌させる。(ただし5分以内)
5. 10ml P3 bufferを加えよく混和して15000rpm, 20min5研遠心機でローターを替えて4℃遠心
6. この間以下の準備をする。
(1)50mlのチューブラックをトレイにセットし、カラムをたてる。遠心が(2) ワットマン紙を適宜湿らせて漏斗にセットし、カラムに載せる。
(3) 終わる5分前にカラムを10ml QBT bufferで平衡化する。
7. 上清を漏斗で濾過しながらカラムにapplyし、gravity flowで落とす。
8. Buffer QC約30mlで2回洗浄する。(gravity flowで落とす。)
9. カラムを別の50ml専用遠心管に立て、15ml bufer QFで溶出する。
10. 室温の2-propanol (isopropanol)を10.5ml 添加し、よく混和して15000 rpm, 30min 5研遠心機で4_遠心
11. ペレットを壊さないよう注意深く上清を吸引。
12. 10-15ml 75% EtOHで洗浄、15000 rpm, 10min 5研遠心機で4_遠心
13. ペレットを壊さないよう注意深く上清を吸引、逆さまにして20-30分乾かす。
14. 0.5-1ml TEでペレットを完全に溶かし、ODを測定して-20℃で保存する。
1. Digestion of pBluescript by EcoRV
take 5g DNA
EcoRV: 6
ベーリンガーSURE CUT B buffer: 6オ;l
+H2O
2. Purification using QIAquick PCR purification kit
3. Addition of T at 3' end
purified DNA, 16-20l
dTTP, 2l (dissolve new tube with 100
l ddH2O)
AmpriTaq, 1
+H2O
70℃, 2hours
4. Purification again using QIAquick PCR purification kit
5. Aliquot 50-100ng each (5l), use for 1 ligation
血小板を遠心してPRPを採取する。
別プロトコール参照
(注)RNAを取る際にはリンパ球のコンタミを防ぐために一番上の層を取るように心がける。
血小板を再度5000rpm, 5min遠心する。
(注)この後血小板を洗浄する手もあるがRNAのdegradationを恐れて今回は施行しない。
上清PPPを捨てる(または-80℃凍結保存する)
血小板ペレットに800のRNA-STAT 60 solutionを加え、ゆっくりpipettingしながらペレットを溶かす。
この状態で-80℃に凍結して保存できる。
室温で5分
160のクロロフォルムを加え激しく転倒混和した後2ー3分放置
Max speed, 4℃で15分遠心
インターフェースを取らないように注意しながら上半分をあらかじめ0.4mlのイソプロパノールを入れておいたtubeに移す。
室温5ー10分
Max speed, 4℃で10分遠心
ペレットが見えればそれがRNAである。(見えないこともある)上清を捨て1mlの75%エタノールで洗浄して遠心、エタノールを捨ててからペレットを乾かす。
DEPC-treated H2Oにresuspendして-80℃で保存する。
一部を取ってアガロースゲル泳動しRNAのqualityを確かめる。
1.Reverse Transcription (First strand synthesis)
量(l) 終濃度
25mM Mg2Cl 4 5mM
5XRT buffer (Gibco) 6
dNTP mix (2.5mM) 8
RNAse inhibitor 1
Reverse Transcriptase 1
Random hexamer or oligo dT 1
total RNA+DEPCH2O 9
合計 30
PCR macine(PCR 9600)を用い、
42℃ 60min
99℃ 5min
5℃ 5min
反応終了後は-20℃で保存
2. PCR reaction
mixture 1
量(l) 終濃度
10X PCR buffer 10
AmpriTaq DNA polymerase or
TAKARA polymerase 1
RT reaction mix + H2O 85
これらをPCR 9600にセットして94℃にheat up
mixture 2
量(l)
Primer 1 (20pmol/l) 2
Primer 2 (20pmol/l) 2
これをmixture 1に加えて以下のPCR反応をスタート
PCR反応(この反応は一例で組み合わせによって変更する場合あり)
94℃ 15sec
60℃ 30-1min
35 cycles
反応後は4℃で保存、"Agarose gel electrophoresis"のプロトコールに従ってPCR productを泳動して確認
(1)DNAの流れている部分のみge1をcutする。
(2)1.5M−NaC1, 0.5M−NaOH buff_で45min,タッパの中でge1をshake。
(3)aspiraterでbuffer吸引
(4)1M−Tris(pH7.0), 1.5M−NaC1 bufferで 45min shake
Whatman 3MM paper 4枚(cut ge1より少し小さく(A))
membrane(Zetaprobeなど、cut gelと同じ大きさ)
Whatman 3MM paper 2枚(大きい奴(B))
paper towelたくさん(cutge1より少し小さく)
(5)x10−SSCで10min shake
(6)200m1, x1−SSCにmembraneとWhatman 3MM paper (A)を浸しておく。
(7) setting
(8)数時間後paper tissuesを交換、x10−SSCを追加。
(1)overnight
(10)自然乾燥、Whatmann paperにはさんでbake(80℃、90min)。
Megaprime DNA labeling kit
アイソトープ8番横置きフリーザーに入っています。
切り出したprobe 25-100ng (1-10lまで)
primer:solution1 5l
ボイル、5min(PCR macineを使うとよい)
megaprime reaction buffer (solution 2) 10
unlabeled dNTP (solution 2a-d): dATP, dGTP, dTTP各4オ;l
reaction buffer (solution2e) 5l
32P-dCTP, 5l
enzyme, solution 3, 2
add water to make up 50l
37℃, 5-10min
stop reaction by adding 2l of 0.5M EDTA
Removal of unlabeled isotope by ProbeQuant -50 Micro Columns
カラムはアイソトープ8番赤い冷蔵庫に入っています。
vortexで中のresinをresuspend
capを1/4ゆるめ、1.5mlエッペンにいれて3000rpm, 1min遠心
新しい1.5mlエッペンにいれて、
ラベルしたsampleを入れる
capが1/4ゆるんでいるのを確認し、3000rpm, 2min遠心
約75のラベルされたprobeがエッペンに回収される
カラムはhotなのでアクリル板コーナーの中で減衰させてから捨てる
QuikHyb Hybridization Solutionはアイソトープ18番奥の冷蔵庫に入っています。
準備
QuikHybは使用前によく振り、65℃に暖めておく
使用量:最低33オ;l/cm2 membrane
membraneが小さければ50mlのtubeに2mlでよい
Hybridization ovenをprewarmしておく65℃
Prehybridization
10-20min
この間にprobeにsalmon sperm DNA(final 100g/ml)をいれボイル、2min(PCR macineを使うとよい)
Hybridization
membraneが小さければ50mlのtubeの中で、
Prehybridization solutionを捨てPrehybridizationと同量の新しいQuikHyb Solutionにボイルしたprobeをいれ、tubeに入れて65℃1hr
membraneが大きければ
Prehybridization solutionから1mlをとり、ボイルしたprobeと混ぜてから戻して65℃1hr